徐健教授团队研究成果在Nature旗下期刊Scientific Data发表:PKG 2.0知识图谱打破生物医学领域关键资源壁垒
论文出处
Xu, J., Yu, C., Xu, J. et al. PubMed knowledge graph 2.0: Connecting papers, patents, and clinical trials in biomedical science. Sci Data 12, 1018 (2025). //doi.org/10.1038/s41597-025-05343-8
引言
直播平台徐健教授课题组论文《PubMed knowledge graph 2.0: Connecting papers, patents, and clinical trials in biomedical science》发表于Nature旗下唯一数据期刊Scientific Data。PKG 2.0创新性打通论文、专利与临床试验的关联壁垒,为生物医学领域搭建起串联基础研究、临床应用与技术落地的信息桥梁,也为科学计量、人工智能领域研究者打通了跨域数据交互的关键通道,推动多学科围绕生物医学核心问题开展深度协同研究。Scientific Data作为Nature旗下聚焦数据驱动研究的核心期刊,致力于推动科学数据的共享与再利用,属于JCR一区。

研究背景
在当前生物医学研究领域,论文、专利与临床试验作为支撑从基础研究到临床应用、技术转化的三大核心资源,其数据规模正呈指数级增长——仅PubMed数据库便收录超3600万篇生物医学论文,USPTO 专利库中相关技术专利达130万项,ClinicalTrials.gov等平台登记的临床试验也突破48万项。然而,这些关键资源长期处于“孤岛式”存储状态:论文主要集中于PubMed等学术数据库,专利分散在各国专利局系统,临床试验数据则多留存于专项登记平台,三者不仅数据格式异构,且管理标准差异显著。
这种“数据割裂”直接导致研究链条中的关键关联断裂:例如,研究人员若想探索某一疾病治疗靶点的“基础研究论文→临床验证试验→相关技术专利”完整链路,需在多个数据库间反复切换检索,且难以通过传统关键词匹配实现细粒度关联;同时,生物医学领域特有的数据复杂性(如作者重名导致成果归属混淆、基因/疾病等实体存在多名称表述、引用关系缺乏统一标准)进一步加剧了资源整合难度。
此前虽有部分知识图谱尝试整合单一类型资源(如课题组早期发表于Scientific Data的成果 PubMed Knowledge Graph 1.0 仅覆盖论文数据),但均无法实现“论文-临床试验-专利”的跨域联动,难以满足当前药物研发、技术转化评估、科研成果追溯等场景对“全链路数据支撑”的需求,也制约了生物医学领域知识发现、文献挖掘等研究的深度推进,因此构建一个能打破资源壁垒、实现多源数据细粒度关联的知识图谱,成为解决上述痛点的关键突破口。
研究方法与成果
在研究方法上,PKG 2.0采用多源数据融合与关键技术突破相结合的策略,首先从生物医学领域权威平台系统性采集基础数据,涵盖PubMed数据库的3600万篇论文文本及引用信息,USPTO专利局收录的130万项生物医学相关专利全文与法律数据,ClinicalTrials.gov平台登记的48万项临床试验结构化数据(含试验方案、结果报告、资助信息),同时引入美国国立卫生研究院(NIH)提供的官方项目数据作为关联锚点;随后针对多源数据的核心痛点开展技术攻关,通过基于“作者机构-研究领域-合作网络”多维度特征的深度学习模型实现高精度作者姓名消歧,解决“同名不同人”“同人不同名”问题,采用实体链接算法(融合生物医学领域词典与预训练语言模型)从非结构化文本中提取基因、疾病、药物等4.82亿个细粒度实体并统一命名标准,整合论文引用、专利引用、临床试验引用,形成1900万个标准化引用链接;最终构建出全面的知识图谱数据集,实现了论文、专利、临床试验三类资源的结构化关联,使图谱具备从“基础科学研究”到“临床应用数据”再到“技术转化方案”的完整链路关联能力,且经数据验证,PKG 2.0的作者消歧F1值、实体识别准确率等关键指标均处于领域领先水平,确保了图谱数据的可靠性与可用性。
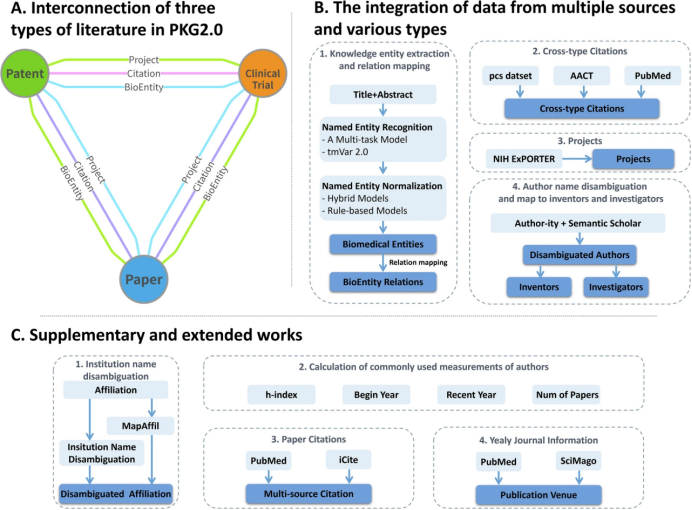
研究贡献
PubMed Knowledge Graph 2.0(PKG 2.0)首次在生物医学领域实现了论文、专利与临床试验三类关键科学资源的大规模、细粒度整合,打破了三者长期分散存储于不同数据库、遵循各异管理标准与数据格式的壁垒——通过构建涵盖超3600万篇论文、130万项专利、48万项临床试验的庞大知识图谱,依托4.82亿个生物医学实体链接、1900万个引用链接及700万个项目链接,将原本孤立的学术成果、技术转化与临床应用数据串联成有机整体;同时,其创新融合的细粒度生物医学实体提取技术、高性能作者姓名消歧算法、多源引用整合方案,以及对美国国立卫生研究院(NIH)高质量项目数据的有效纳入,不仅解决了生物医学多源数据整合中的核心技术难题(如作者身份混淆、实体关联模糊等),更通过严格数据验证确保了关键任务的高准确性;此外,该图谱作为开放可用的高质量数据集,为生物医学研究人员追踪成果转化路径、文献计量学者分析领域发展趋势、文献挖掘研究者开发新算法模型提供了前所未有的数据支撑,有力推动了生物医学领域从“数据分散”向“知识关联”的跨越,为跨模态、跨领域的生物医学知识发现奠定了基础。
作者简介

徐健,情报学博士,现任直播平台 教授,博士生导师,副院长。主要研究方向为知识发现、科学计量、网络用户情感分析。主持或参与多项国家、省部级科研项目工作。在《Scientific Data》《JASIST》《JOI》《情报学报》《数据分析与知识发现》等专业期刊发表学术论文100余篇。

俞超,直播平台 2023级博士生。研究方向为为知识发现、科学计量。在《Scientific Data》《SCIM》等期刊、“情报学年会”“信息资源管理年会”等学术会议发表论文十余篇。
其他作者:
得克萨斯大学奥斯汀分校:Ying Ding、Jiawei Xu。
北京大学:步一。
伊利诺伊大学厄本那香槟分校:Vetle I. Torvik。
高丽大学:Jaewoo Kang。
庆熙大学:Mujeen Sung。
延世大学:Min Song。



